ロジスティック回帰の小ネタ
小ネタ①:ロジスティック回帰は集計値を用いても同じ結果となる
tmp <- epitools::expand.table(Titanic) library(tidyverse) dat_table <- tmp %>% group_by(Class, Sex, Age) %>% summarise("Yes" = sum(Survived == "Yes"), "No" = sum(Survived == "No"))
集計値
> summary(glm(cbind(Yes, No) ~ ., dat_table, family = binomial("logit")))$coef Estimate Std. Error z value Pr(>|z|) (Intercept) 0.6853195 0.2729943 2.510380 1.206013e-02 Class2nd -1.0180950 0.1959976 -5.194427 2.053519e-07 Class3rd -1.7777622 0.1715666 -10.361935 3.694112e-25 ClassCrew -0.8576762 0.1573389 -5.451138 5.004844e-08 SexFemale 2.4200603 0.1404101 17.235655 1.434207e-66 AgeAdult -1.0615424 0.2440257 -4.350125 1.360598e-05
生データ
> summary(glm(Survived ~ ., tmp, family = binomial("logit")))$coef Estimate Std. Error z value Pr(>|z|) (Intercept) 0.6853195 0.2729934 2.510388 1.205986e-02 Class2nd -1.0180950 0.1959969 -5.194443 2.053331e-07 Class3rd -1.7777622 0.1715657 -10.361993 3.691891e-25 ClassCrew -0.8576762 0.1573387 -5.451147 5.004592e-08 SexFemale 2.4200603 0.1404093 17.235750 1.431830e-66 AgeAdult -1.0615424 0.2440247 -4.350143 1.360490e-05
標準誤差がわずかに異なるものの、回帰係数は一致。
小ネタ②:ロジスティック回帰とロジットに対する線形回帰は異なる
set.seed(123) n <- 10000 x1 <- sample(c("A", "B"), n, replace = T) x1_A <- ifelse(x1 == "A", 1, 0) x1_B <- ifelse(x1 == "B", 1, 0) x2 <- sample(c("X", "Y", "Z"), n, replace = T) x2_X <- ifelse(x2 == "X", 1, 0) x2_Y <- ifelse(x2 == "Y", 1, 0) x2_Z <- ifelse(x2 == "Z", 1, 0) b0 <- 0 b1_B <- 1.0 b2_Y <- 1.5 b2_Z <- 3.0 l <- b0 + x1_B * b1_B + x2_Y * b2_Y + x2_Z * b2_Z p <- exp(l) / (1 + exp(l)) dat <- data.frame( y = rbinom(n, 1, p), x1 = x1, x2 = x2 ) tmp <- dat %>% group_by(x1, x2) %>% summarise("Yes" = sum(y == 1), "No" = sum(y == 0)) %>% mutate(p = Yes / (Yes + No)) %>% mutate(logit = log(p / (1-p)))
> summary(glm(y ~ x1 + x2, dat, family = binomial("logit")))$coef Estimate Std. Error z value Pr(>|z|) (Intercept) 0.003870327 0.04524904 0.0855339 9.318369e-01 x1B 1.005336861 0.05966550 16.8495494 1.057171e-63 x2Y 1.548350891 0.06500045 23.8206168 2.042443e-125 x2Z 3.016559809 0.10570629 28.5371830 4.051563e-179
> summary(lm(logit ~ x1 + x2, tmp))$coef Estimate Std. Error t value Pr(>|t|) (Intercept) -0.03945677 0.09246599 -0.4267166 0.711129289 x1B 1.08845178 0.09246599 11.7713743 0.007139621 x2Y 1.55366958 0.11324725 13.7192702 0.005271007 x2Z 3.08631608 0.11324725 27.2529016 0.001343688
回帰係数は概ね一致するものの標準誤差に大きな違いがあり、結果的にP値は大きく異なる。
RとPythonの結果を一致させたい
背景
通常、私はデータを分析する際には主にRを使用します。しかし分析結果を既存のシステムに投入するなど実装を考えた場合には、Rではなく別の言語を求められることもあると思います。 今回、RとPythonのそれぞれでGLMを実行したときに結果が一致するのかを検証する機会があったため、その内容を備忘がてら紹介しておきます。
RからPythonを呼び出す
今回の検証では、RからPythonを呼び出すためのライブラリreticulateを使用しました。RStudioからR Notebookとして使えば、chunkごとにRとPythonを切り替えることができて大変便利です。
なおMacユーザーでPython環境としてAnacondaをインストールしている場合、.Rprofileに以下を記載しておくことでAnaconda配下のPythonを呼び出すことができます。
Sys.setenv(PATH = paste("/anaconda3/bin", Sys.getenv("PATH"), sep=":"))
では早速reticulateをインストールし、Pythonを呼び出せるか試してみましょう。
install.packages("reticulate") library(reticulate)
> system("python --version") Python 3.6.4 :: Anaconda custom (64-bit)
このような表示が出れば成功です。
線形回帰
まずはじめに、irisのデータを用いて線形回帰をかけてみます。Rでは以下のように書くことができます。
> glm(Sepal.Length ~ Petal.Length, data = iris, family = gaussian) Call: glm(formula = Sepal.Length ~ Petal.Length, family = gaussian, data = iris) Coefficients: (Intercept) Petal.Length 4.3066 0.4089 Degrees of Freedom: 149 Total (i.e. Null); 148 Residual Null Deviance: 102.2 Residual Deviance: 24.53 AIC: 160
上記をPythonのstatsmodelsを使って同じように書いてみましょう。irisのデータは事前にRから書き出しておいたものを使用することとし、pandasのread_csvで読み込みます。そのデータをstatsmodelsのGLMに渡します。
import statsmodels.api as sm import pandas as pd iris = pd.read_csv("/YourDirectory/iris.csv") res_iris = sm.GLM(iris['Sepal.Length'], iris['Petal.Length'], family = sm.families.Gaussian()).fit() print(res_iris.summary()) Generalized Linear Model Regression Results ============================================================================== Dep. Variable: Sepal.Length No. Observations: 150 Model: GLM Df Residuals: 149 Model Family: Gaussian Df Model: 0 Link Function: identity Scale: 3.521367262403398 Method: IRLS Log-Likelihood: -306.75 Date: Mon, 04 Jun 2018 Deviance: 524.68 Time: 18:30:59 Pearson chi2: 525. No. Iterations: 2 ================================================================================ coef std err z P>|z| [0.025 0.975] -------------------------------------------------------------------------------- Petal.Length 1.3489 0.037 36.530 0.000 1.277 1.421 ================================================================================
ひとまず分析を実行することはできたようですが、Rの結果とは違いますね。出力されたテーブルの下部にある「coef」を見てみると、切片が出力されていません。statsmodelsはデフォルトでは切片が入らないようです。
statsmodelsで切片を追加するにはadd_constantを使います。以下のように修正しましょう。
import statsmodels.api as sm import pandas as pd iris = pd.read_csv("/YourDirectory/iris.csv") res_iris_2 = sm.GLM(iris['Sepal.Length'], sm.add_constant(iris['Petal.Length']), family = sm.families.Gaussian()).fit() print(res_iris_2.summary()) Generalized Linear Model Regression Results =============================================================================== Dep. Variable: Sepal.Length No. Observations: 150 Model: GLM Df Residuals: 148 Model Family: Gaussian Df Model: 1 Link Function: identity Scale: 0.16570968760697133 Method: IRLS Log-Likelihood: -77.020 Date: Thu, 31 May 2018 Deviance: 24.525 Time: 13:37:26 Pearson chi2: 24.5 No. Iterations: 2 ================================================================================ coef std err z P>|z| [0.025 0.975] -------------------------------------------------------------------------------- const 4.3066 0.078 54.939 0.000 4.153 4.460 Petal.Length 0.4089 0.019 21.646 0.000 0.372 0.446 ================================================================================
無事、Rの結果と一致しました。
ロジスティック回帰
続いてTitanicのデータを用いてロジスティック回帰をかけてみます。Titanicのデータは下記のコードによりdataframeに加工し、iris同様に書き出しておきました。
tmp <- data.frame(Titanic) titanic <- data.frame(Class = rep(tmp$Class, tmp$Freq), Sex = rep(tmp$Sex, tmp$Freq), Age = rep(tmp$Age, tmp$Freq), Survived = rep(tmp$Survived, tmp$Freq))
Rでは以下のように書けます。
> glm(Survived ~ ., data = titanic, family = binomial(link = "logit")) Call: glm(formula = Survived ~ ., family = binomial(link = "logit"), data = titanic) Coefficients: (Intercept) Class2nd Class3rd ClassCrew SexMale AgeChild 2.0438 -1.0181 -1.7778 -0.8577 -2.4201 1.0615 Degrees of Freedom: 2200 Total (i.e. Null); 2195 Residual Null Deviance: 2769 Residual Deviance: 2210 AIC: 2222
これをPythonで実行してみましょう。ここではRのformulaと同じように指定するためにstatsmodels.formula.apiを使用します。
import statsmodels.api as sm import statsmodels.formula.api as smf import pandas as pd import numpy as np dat = pd.read_csv("/YourDirectory/titanic.csv") dat = dat.drop('Unnamed: 0', axis = 1) titanic_formula = "Survived ~ Class + Sex + Age" glm_bin = smf.glm(formula = titanic_formula, data = dat, family = sm.families.Binomial(sm.families.links.logit)).fit() print(glm_bin.summary()) Generalized Linear Model Regression Results ============================================================================================= Dep. Variable: ['Survived[No]', 'Survived[Yes]'] No. Observations: 2201 Model: GLM Df Residuals: 2195 Model Family: Binomial Df Model: 5 Link Function: logit Scale: 1.0 Method: IRLS Log-Likelihood: -1105.0 Date: Mon, 04 Jun 2018 Deviance: 2210.1 Time: 16:58:29 Pearson chi2: 2.25e+03 No. Iterations: 5 ================================================================================= coef std err z P>|z| [0.025 0.975] --------------------------------------------------------------------------------- Intercept -2.0438 0.168 -12.171 0.000 -2.373 -1.715 Class[T.2nd] 1.0181 0.196 5.194 0.000 0.634 1.402 Class[T.3rd] 1.7778 0.172 10.362 0.000 1.441 2.114 Class[T.Crew] 0.8577 0.157 5.451 0.000 0.549 1.166 Sex[T.Male] 2.4201 0.140 17.236 0.000 2.145 2.695 Age[T.Child] -1.0615 0.244 -4.350 0.000 -1.540 -0.583 =================================================================================
やはり、結果が異なります。よく見ると、カテゴリカル変数についてRではFemaleとAdultが推定されているのに対して、PythonではMaleとChildが推定されていることがわかります。どうやら参照列(Reference)が異なるようです。
Rの方で、Referenceを変更して確認してみましょう。
titanic$Sex <- relevel(titanic$Sex, ref = "Female") titanic$Age <- relevel(titanic$Age, ref = "Adult") > glm(Survived ~ ., data = titanic, family = binomial(link = "logit")) Call: glm(formula = Survived ~ ., family = binomial(link = "logit"), data = titanic) Coefficients: (Intercept) Class2nd Class3rd ClassCrew SexMale AgeChild 2.0438 -1.0181 -1.7778 -0.8577 -2.4201 1.0615 Degrees of Freedom: 2200 Total (i.e. Null); 2195 Residual Null Deviance: 2769 Residual Deviance: 2210 AIC: 2222
これで結果が近くなりましたが、まだ一致していません。回帰係数の絶対値は同じとなりましたが符号が異なっています。目的変数であるYesとNoという文字列が、RとPythonで別々に解釈された可能性があります。
今度はPython側でYesとNoの定義を修正してみましょう。
import statsmodels.api as sm import statsmodels.formula.api as smf import pandas as pd import numpy as np dat = pd.read_csv("/YourDirectory/titanic.csv") dat = dat.drop('Unnamed: 0', axis = 1) dat['Survived'] = dat['Survived'].str.replace('No', '0') dat['Survived'] = dat['Survived'].str.replace('Yes', '1') dat['Survived'] = dat['Survived'].astype(np.int64) titanic_formula = "Survived ~ Class + Sex + Age" glm_bin_2 = smf.glm(formula = titanic_formula, data = dat, family=sm.families.Binomial(sm.families.links.logit)).fit() print(glm_bin_2.summary()) Generalized Linear Model Regression Results ============================================================================== Dep. Variable: Survived No. Observations: 2201 Model: GLM Df Residuals: 2195 Model Family: Binomial Df Model: 5 Link Function: logit Scale: 1.0 Method: IRLS Log-Likelihood: -1105.0 Date: Mon, 04 Jun 2018 Deviance: 2210.1 Time: 17:03:20 Pearson chi2: 2.25e+03 No. Iterations: 5 ================================================================================= coef std err z P>|z| [0.025 0.975] --------------------------------------------------------------------------------- Intercept 2.0438 0.168 12.171 0.000 1.715 2.373 Class[T.2nd] -1.0181 0.196 -5.194 0.000 -1.402 -0.634 Class[T.3rd] -1.7778 0.172 -10.362 0.000 -2.114 -1.441 Class[T.Crew] -0.8577 0.157 -5.451 0.000 -1.166 -0.549 Sex[T.Male] -2.4201 0.140 -17.236 0.000 -2.695 -2.145 Age[T.Child] 1.0615 0.244 4.350 0.000 0.583 1.540 =================================================================================
これで一致しました。
終わりに
今回見てきたように、同一かつシンプルなモデルであっても、言語の仕様によって出力は容易に変わります。異なる結果が得られたときに大慌てしなくて済むよう、各言語や関数についての仕様を把握しておき、本当に異なる結果となっているのかをチェックするというのが大切です。特にモデルの結果を実システムに組み込む際にはエンジニアによるリファクタリングが行われることも多いでしょうから、エンジニアがハマりそうなポイントを押さえておくと役立ちそうですね。
Stanを使って変数選択したい
背景
Stanを使ってモデリングをしている時に不満を感じる点として、変数選択が難しいということが挙げられます。もともと私自身は、例えばStepwiseやLassoなどを用いた"機械的な"変数選択があまり好きではない1のですが、それでも分析を効率的に進める上でそれらの手法に頼りたくなることがあります。
そういったときにglmを用いているのであればstep関数により容易に変数選択が可能なのですが、Stanではそうもいきません。何か良い方法はないかと探していたところ、StanのGithubレポジトリに{projpred}というそれっぽいlibraryを見つけたので、紹介がてら変数の選択精度を実験してみます2。
データ準備
ライブラリの読み込み
今回の分析では{rstan}の代わりに{rstanarm}というlibraryを使用します。{rstanarm}はRのglmと同じようなシンタックスのままでモデルをベイズ化することが可能で、例えば{rstanarm}のvignetteでは以下のようなスニペットが紹介されています(一部改変あり)3。
library(rstanarm) data("womensrole", package = "HSAUR3") womensrole$total <- womensrole$agree + womensrole$disagree womensrole_bglm_1 <- stan_glm(cbind(agree, disagree) ~ education + gender, data = womensrole, family = binomial(link = "logit"), prior = student_t(df = 7), prior_intercept = student_t(df = 7), chains = 4, cores = 1, seed = 123)
> womensrole_bglm_1 stan_glm family: binomial [logit] formula: cbind(agree, disagree) ~ education + gender observations: 42 predictors: 3 ------ Median MAD_SD (Intercept) 2.5 0.2 education -0.3 0.0 genderFemale 0.0 0.1 Sample avg. posterior predictive distribution of y: Median MAD_SD mean_PPD 24.3 0.8 ------ For info on the priors used see help('prior_summary.stanreg').
数字の意味は一旦置いておきますが、今回はこちらのlibraryを使用しながら進めていきます。
続いて他のlibraryを読み込みます。この辺りはvignetteそのままです。
library(projpred) library(ggplot2) library(bayesplot) theme_set(theme_bw()) options(mc.cores = parallel::detectCores())
シミュレーションデータの作成
実験を進めるにあたりシミュレーションデータを作成します。もちろんlibraryに付属のデータセットを使っても良いのですが、そのまま同じことをするのも面白くないので以下のように複数のデータを作成して結果を比較してみましょう:
- 説明変数の数が少なく(20)、連続変数のみ
- 説明変数の数が多く(100)、連続変数のみ
- 説明変数の数が少なく(20)、ダミー変数を含む
- 説明変数の数が多く(100)、ダミー変数を含む
見てわかる通りで、説明変数の数とダミー変数の有無によって4パターンを用意します。なぜこのパターンにしたかというと、単純にvignetteを見ていて連続変数ばかりだなと思ったのと、サンプルデータの説明変数の数が20ぐらいで、これなら機械的な選択に頼らなくても自力でどうにかできそうだな、と思ったためです。
各パターンについて特にひねりもなくデータを作成します。なお真のモデルは1と2、3と4で同一のものとしました。したがって2と4の状況は、1と3それぞれに比べて「不要なデータのみが追加された状態」での変数選択という状況になります。
以下のように各パラメータを設定します。
set.seed(1) N <- 100 # number of observations xn_1 <- 20 # number of variables for pattern 1 xn_2 <- 100 # number of variables for pattern 2 b1 <- 0.5 # regression coefficient of X[, 1] b2 <- 0.8 # regression coefficient of X[, 2] var_e <- 1 # residual variance
パターン1 & 2について、まず乱数によりXを生成し、そのうちの2列を使ってYを作成します。またその2列を含む20列および全列を各パターンの説明変数とします。
X <- matrix(rnorm(N * xn_2, 0, 1), nrow = N, ncol = xn_2) Y <- 1.0 + X[, 1] * b1 + X[, 2] * b2 + rnorm(N, 0, var_e) X1 <- X[, 1:xn_1] X2 <- X[, 1:xn_2] dat1 <- data.frame("Y" = Y, "X" = I(X1)) dat2 <- data.frame("Y" = Y, "X" = I(X2))
データセットを作成する際にX1をI()で渡すことで、X1全体を"X"として再定義できます。すると以下のようなformulaが実行可能になります。
> lm(Y ~ X, dat1) Call: lm(formula = Y ~ X, data = dat1) Coefficients: (Intercept) X1 X2 X3 X4 X5 X6 1.038393 0.422365 0.824111 0.006940 -0.064897 0.060024 -0.004623 X7 X8 X9 X10 X11 X12 X13 -0.096471 0.040521 0.123807 -0.051311 0.125116 0.009435 0.014924 X14 X15 X16 X17 X18 X19 X20 0.065920 -0.037135 -0.110772 -0.015537 -0.003969 -0.136834 0.095512
もちろんそのまま渡して.で指定するのでも構いません。
tmp <- data.frame("Y" = Y, "X" = X1)
> lm(Y ~ ., tmp) Call: lm(formula = Y ~ ., data = tmp) Coefficients: (Intercept) X.1 X.2 X.3 X.4 X.5 X.6 1.038393 0.422365 0.824111 0.006940 -0.064897 0.060024 -0.004623 X.7 X.8 X.9 X.10 X.11 X.12 X.13 -0.096471 0.040521 0.123807 -0.051311 0.125116 0.009435 0.014924 X.14 X.15 X.16 X.17 X.18 X.19 X.20 0.065920 -0.037135 -0.110772 -0.015537 -0.003969 -0.136834 0.095512
続いてパターン3 & 4についても基本的に同じ流れでデータを作成しますが、一部にダミー変数を含めます。
Z <- matrix(rnorm(N * xn_2, 0, 1), nrow = N, ncol = xn_2) Z[, seq(1, 100, 10)] <- if_else(Z[, seq(1, 100, 10)] > 0, 1, 0) Y <- 1.0 + Z[, 1] * b1 + Z[, 2] * b2 + rnorm(N, 0, var_e) X3 <- Z[, 1:xn_1] X4 <- Z[, 1:xn_2] dat3 <- data.frame("Y" = Y, "X" = I(X3)) dat4 <- data.frame("Y" = Y, "X" = I(X4))
フィッティング
stan_glmによるフィッティング
では、これらのデータを用いてフィッティングをしてみましょう。スクリプトは以下のようになります:
n <- N D <- xn_1 p0 <- 5 # prior guess for the number of relevant variables # scale for tau (notice that stan_glm will automatically scale this by sigma) tau0 <- p0 / (D-p0) * 1/sqrt(n) prior_coeff <- hs(global_scale = tau0, slab_scale = 1) # regularized horseshoe prior fit1 <- stan_glm(Y ~ X, family = gaussian(), data = dat1, prior = prior_coeff, seed = 777, chains = 4, iter = 2000)
D <- xn_2 p0 <- 5 tau0 <- p0/(D-p0) * 1/sqrt(n) prior_coeff <- hs(global_scale = tau0, slab_scale = 1) fit2 <- stan_glm(Y ~ X, family = gaussian(), data = dat2, prior = prior_coeff, seed = 777, chains = 4, iter = 2000)
D <- xn_1 p0 <- 5 tau0 <- p0 / (D-p0) * 1/sqrt(n) prior_coeff <- hs(global_scale = tau0, slab_scale = 1) fit3 <- stan_glm(Y ~ X, family = gaussian(), data = dat3, prior = prior_coeff, seed = 777, chains = 4, iter = 2000)
D <- xn_2 p0 <- 5 tau0 <- p0 / (D-p0) * 1/sqrt(n) prior_coeff <- hs(global_scale = tau0, slab_scale = 1) fit4 <- stan_glm(Y ~ X, family = gaussian(), data = dat4, prior = prior_coeff, seed = 777, chains = 4, iter = 2000)
実行画面は省略します。
結果の確認
無事にフィッティングが終わったのでまずは結果を確認しましょう。fit1オブジェクトには多くの情報が格納されていますが、以下のようにパラメータの分布を確認します(結果は一部抜粋)。
> fit1$stan_summary mean se_mean sd 2.5% 10% 25% 50% 75% 90% 97.5% n_eff Rhat (Intercept) 1.026278e+00 0.0012818960 0.08107422 0.87096060 0.92283578 9.695387e-01 1.025776e+00 1.083455e+00 1.12962235 1.18570039 4000.000 1.0002199 X1 3.908290e-01 0.0016254360 0.09798225 0.19341915 0.26638997 3.278100e-01 3.940279e-01 4.542220e-01 0.51236797 0.57793402 3633.750 0.9999048 X2 8.240328e-01 0.0013608445 0.08516982 0.65884688 0.71552972 7.672683e-01 8.238806e-01 8.816342e-01 0.93310548 0.98857084 3917.008 0.9995082 X3 -1.059357e-03 0.0005388889 0.03408233 -0.08025937 -0.03484370 -1.106505e-02 -1.177711e-04 1.032889e-02 0.03180827 0.07368474 4000.000 1.0005552 X4 -1.872770e-02 0.0007089570 0.04483838 -0.13979248 -0.07768171 -3.228712e-02 -3.977780e-03 3.213492e-03 0.01638515 0.04656703 4000.000 0.9998376 X5 2.138502e-02 0.0006738661 0.04261903 -0.03511247 -0.01201396 -1.858049e-03 6.263328e-03 3.500771e-02 0.07857940 0.13847975 4000.000 0.9993905# ︙ # 以下省略 # ︙
この結果を見るとX1とX2で概ね設定通りの値が得られているようですね。
ではここから{projpred}による変数選択に取り掛かりましょう。スクリプトは以下のようになります:
sel1 <- varsel(fit1, method = 'forward')
> sel1$varsel$vind # variables ordered as they enter during the search X2 X1 X20 X16 X19 X11 X5 X14 X18 X4 X7 X9 X8 X17 X10 X15 X13 X12 X3 X6 2 1 20 16 19 11 5 14 18 4 7 9 8 17 10 15 13 12 3 6
おおっ!ちゃんと1列目と2列目が選ばれていますね!下記のプロットを見ても、3番目以降の変数はモデルの精度に対して寄与していないことがわかると思います。なおここでelpdはexpected log predictive densityを指しますが、ちょっと情報量規準辺りの話はうかつに解説できないのでvignetteを引用するに留めておきます。
The loo method for stanreg objects provides an interface to the loo package for approximate leaveone-out cross-validation (LOO). The LOO Information Criterion (LOOIC) has the same purpose as the Akaike Information Criterion (AIC) that is used by frequentists. Both are intended to estimate the expected log predictive density (ELPD) for a new dataset.
varsel_plot(sel1, stats=c('elpd', 'rmse'))

各パラメータの分布についても見てみましょう。上位5個に選ばれた説明変数に限定してプロットしてみます。
# Visualise the three most relevant variables in the full model mcmc_areas(as.matrix(sel1), pars = names(sel1$varsel$vind[1:5])) + coord_cartesian(xlim = c(-2, 2))

効果のなかった変数は0を中心に集中して分布していることがわかります。
残りの各パターンについても同様に見ていきますが、個別に確認するのは面倒なので一度にまとめてしまします。
sel2 <- varsel(fit2, method = 'forward') sel3 <- varsel(fit3, method = 'forward') sel4 <- varsel(fit4, method = 'forward')
> sel2$varsel$vind X2 X1 X87 X24 X83 X71 X89 X18 X95 X85 X4 X67 X19 X16 X60 X56 X55 X53 X42 X20 2 1 87 24 83 71 89 18 95 85 4 67 19 16 60 56 55 53 42 20 > sel3$varsel$vind X2 X14 X4 X1 X18 X5 X16 X3 X9 X15 X19 X20 X6 X8 X17 X11 X7 X12 X13 X10 2 14 4 1 18 5 16 3 9 15 19 20 6 8 17 11 7 12 13 10 > sel4$varsel$vind X2 X90 X74 X45 X64 X14 X25 X52 X60 X4 X20 X16 X22 X1 X63 X41 X9 X26 X53 X5 2 90 74 45 64 14 25 52 60 4 20 16 22 1 63 41 9 26 53 5
むむ。。。
いずれのパターンでも2列目はちゃんと選ばれていますが、パターン3と4では1列目が選ばれていませんね。 プロットも見てみましょう。
varsel_plot(sel2, stats=c('elpd', 'rmse')) varsel_plot(sel3, stats=c('elpd', 'rmse')) varsel_plot(sel4, stats=c('elpd', 'rmse'))

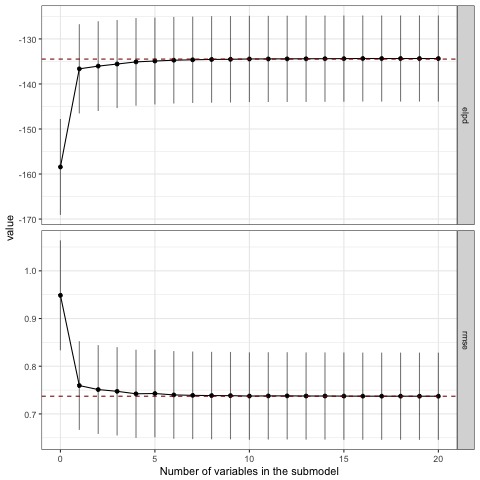
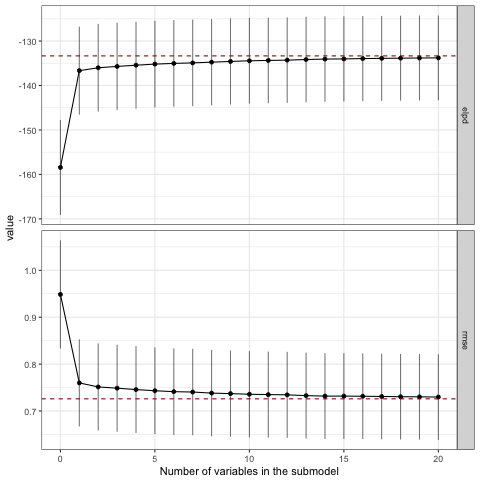
正しい変数セットが選ばれていないので当然ですが、2変数目ですでにフルモデルにおけるelpdやrmseと接するようになっており、モデルの精度改善に貢献しているのは1変数目だけという結果になっています。
パラメータの分布を見ても、パターン1と比較するといずれも分布の裾が広くなっています。さらにパターン3では正の効果を受けたためか右に裾を引く形とはなっているものの、効果のない他の変数とほとんど変わらない分布となってしまいました。ダミー変数はパラメータの推定が難しいようです。
mcmc_areas(as.matrix(sel2), pars = names(sel2$varsel$vind[1:5])) + coord_cartesian(xlim = c(-2, 2)) mcmc_areas(as.matrix(sel3), pars = names(sel3$varsel$vind[1:5])) + coord_cartesian(xlim = c(-2, 2)) mcmc_areas(as.matrix(sel4), pars = names(sel4$varsel$vind[1:5])) + coord_cartesian(xlim = c(-2, 2))



追試
では、ダミー変数が含まれる場合に真のパラメータを捉えることができる確率としてはどの程度になるのでしょうか。パターン3を100回繰り返し、そのうち何回真の変数セットを選択できるか確認してみましょう。
n <- 100 t <- 100 D <- 20 p0 <- 5 b1 <- 0.5 b2 <- 0.8 tau0 <- p0 / (D-p0) * 1/sqrt(n) out <- matrix(0, t, 2) for (i in 1:t) { x <- matrix(rnorm(n * D, 0, 1), nrow = n) x[, seq(1, D, 5)] <- ifelse(x[, seq(1, D, 5)] > 0, 1, 0) y <- 1.5 + x[, 1] * b1 + x[, 2] * b2 + rnorm(n, 0, 1) dat <- data.frame("y" = y, "x" = I(x)) prior_coeff <- hs(global_scale = tau0, slab_scale = 1) fit0 <- stan_glm(y ~ x, family = gaussian(), data = dat, prior = prior_coeff, chains = 4, iter = 2000) sel0 <- varsel(fit0) out[i, ] <- names(sel0$varsel$vind[1:2]) }
> table(out[, 1]) x2 100 > table(out[, 2]) x1 x10 x11 x12 x13 x14 x15 x17 x18 x19 x20 x3 x4 x5 x6 x7 x9 55 2 2 3 3 1 1 5 5 1 1 2 7 4 1 4 3
なんと、連続変数である2列目の選択確率は100%である一方、ダミー変数である1列目は約半数しか選択されないという結果になりました。もちろん回帰係数の大小にも影響されるのでしょうが、結構衝撃的な結果に思えます。
終わりに
これまでの実験の結果をまとめると以下のようになりそうです:
- 連続変数であれば説明変数を20から100に増やしても大きな問題なく変数を選択できそう
- ダミー変数の変数選択は難しく、今回の条件の場合、半分の確率で失敗する
- ダミー変数はパラメータの推定も難しい
実際のデータ分析の現場では真のパラメータを予め知ることができないため、変数選択の結果を受け入れざるを得ないことがあるかと思います。しかし今回の実験の結果からは「真の変数セットを選択できる確率は半分程度しかない」ことが示されており、機械的な変数の取捨選択がどれほど危険であるかを教えてくれているのではないでしょうか。
とは言え今回実験に使用した{projpred}は便利なlibraryであると思いますので、これから活用しつつも引き続き変数選択の方法について探していこうと思います。
Ad-Stock効果を推定しつつ回帰を回したい⑤
背景
しつこいようですが、Marketing Mix Modeling(MMM)の話題です。
先日、こんな面白い論文を見つけました。 GoogleのResearcherによるMMMの論文(彼らはMedia Mix Modelingと呼んでいます)なのですが、ヒルの式を用いて広告のShape効果(Carveture効果)を推定するということをやっています。ここでShape効果・carveture効果とは、メディアの露出量に対する目的変数の反応を示す曲線を指すようで、ヒルの式とは:
$$ H(x; K, S) = \frac{1}{1 + (\frac{x}{K})^{-S}} $$
であり、$K > 0$や$S > 0$となるパラメータによってLogやSigmoidの形状を表現することができるようです。
ヒルの式によってxがどのような形状となるか、実際に確認してみましょう。まずはヒルの式を以下のように定義します。
hill_transformation <- function(x, k, s) { 1 / (1 + (x / k)^-s) }
続いて、パラメータとなるkとsをそれぞれ以下のように設定した場合をプロットしてみましょう。なおこの数値は論文から拝借しています。
x <- seq(0, 1, by = 0.05) plot(x, hill_transformation(x, 0.4, 4), type = "l", col = 2, xlim = c(0, 1), ylim = c(0, 1), ylab = "Hill Transformed Value") par(new = T) plot(x, hill_transformation(x, 0.4, 1), type = "l", col = 3, xlim = c(0, 1), ylim = c(0, 1), axes = F, ylab = "") par(new = T) plot(x, hill_transformation(x, 0.8, 4), type = "l", col = 4, xlim = c(0, 1), ylim = c(0, 1), axes = F, ylab = "") par(new = T) plot(x, hill_transformation(x, 0.8, 1), type = "l", col = 5, xlim = c(0, 1), ylim = c(0, 1), axes = F, ylab = "") legend("topleft", legend = c("k = 0.4, s = 4", "k = 0.4, s = 1", "k = 0.8, s = 4", "k = 0.8, s = 1"), col = c(2:5), lty = rep(1, 4))
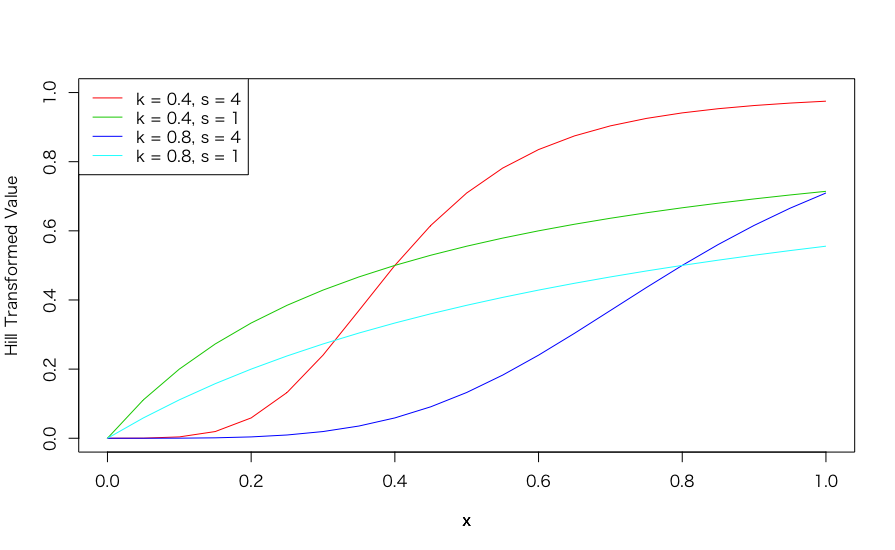
このように、パラメータを変更することで元の値を柔軟に変換することが可能です。これまで私が書いてきた記事では、いずれも広告効果(回帰係数)そのものを推定する方法にばかり注目しており、説明変数の"効き方"については触れてきませんでした。これではちょっと検討が足りないと言われても仕方ありません。
というわけで本エントリーでは、ヒルの式により変換したXを用いてシミュレーションデータを発生させ、元のXから変数変換のためのパラメータを推定できるかを検証したいと思います。なお元論文ではAd-Stock効果としてGeometric Ad-Stock:
$$ GA(x_{t}; \alpha, L) = \frac{\sum_{l = 0}^{L}x_{t-l}\alpha^{l}}{\sum_{l = 0}^{L}\alpha^{l}} $$
を用いていますが、今回の検証ではAd-Stock効果をみておらず、説明変数の生の値を変換しています1。
ライブラリの読み込み
今回の分析でも{rstan}を使用します。
library(tidyverse) library(ggplot2) library(rstan) options(mc.cores = parallel::detectCores()) rstan_options(auto_write = TRUE)
シミュレーションデータの生成
分析用のデータ生成ですが、まず説明変数X1をrunifにより作成します。次に与えたパラメータからヒルの式を用いて変換し、目的変数yを作成します。
## データ生成用の関数を定義 simulate_y <- function(pars) { n <- pars[1] # num of observation mu <- pars[2] # intercept var_e <- pars[3] # residual variance beta_01 <- pars[4] # regression coefficient of X1 to be esitmated # lambda_01 <- pars[5] # decay rate of Ad-Stock effect of X1 k_01 <- pars[6] # k for X1 s_01 <- pars[7] # s for X1 X_01_raw <- runif(n) X_01_conv <- hill_transformation(X_01_raw, k_01, s_01) error <- rnorm(n, 0, sqrt(var_e)) y <- mu + beta_01 * X_01_conv + error dat <- data.frame( "Y" = y, "X_01" = X_01_raw, "X_01_conv" = X_01_conv ) return(na.omit(dat)) } ## データ生成 set.seed(123) pars <- c(100, 5, 0.1, 0.8, 0.7, 0.4, 4) dat <- simulate_y(pars)
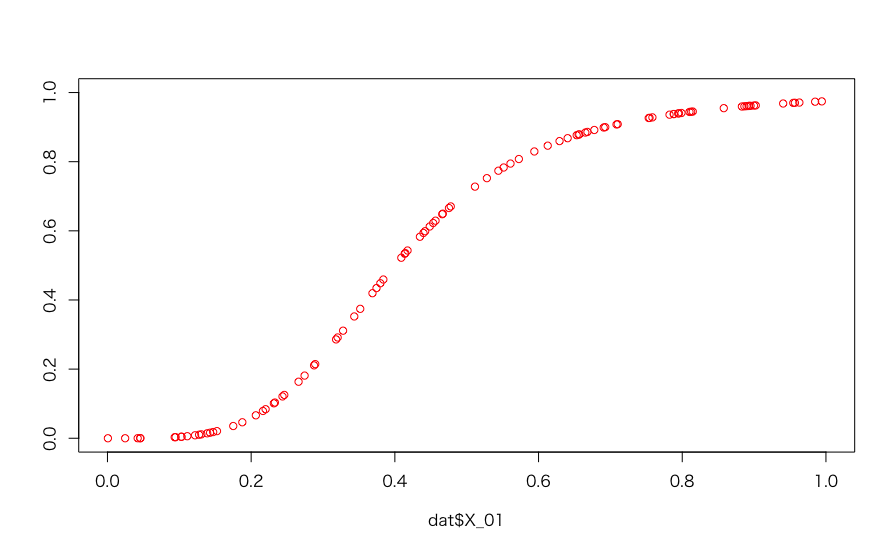
X1について、
- 推定したい広告効果 ⇒
0.8 - ヒルの式の
kおよびs⇒0.4および4
と指定しており、これらのパラメータを元のX_01から推定することが狙いです。ちなみに今回のパラメータではX_01と変換後のX_01は以下のようになります:
plot(dat$X_01, dat$X_01_conv, col = 2, xlim = c(0, 1), ylim = c(0, 1), ylab = "")
optimによるフィッティング
手始めにoptimを使ってフィッティングしてみましょう。これまでの記事でも度々optimを使ってきましたが、なかなか精度良く推定することが可能です。ただしAICを計算するのが面倒なのでここではbetaを直接推定しないことにして、lmを使います。よって推定対象はk_01とs_01の2つだけです。
return_AIC <- function(param, dat) { k_01 <- param[1] s_01 <- param[2] dat$X_01_conv <- hill_transformation(dat$X_01, k_01, s_01) AIC(lm(Y ~ X_01_conv, dat)) }
### 適当な数値を入れてみる > return_AIC(rep(0.5, 2), dat) [1] 60.47417
ちゃんと動くようなのでフィッティングしてみましょう。
param <- rep(1, 2) res_optim <- optim(par = optim(par = param, fn = return_AIC, dat = dat)$par, fn = return_AIC, dat = dat)
パラメータを確認してみると:
true_par <- c(0.8, 0.4, 4) dat$X_01_conv_opt <- hill_transformation(dat$X_01, res_optim$par[1], res_optim$par[2]) optim_par <- c(coef(lm(Y ~ X_01_conv_opt, dat))[2], res_optim$par)
> print(cbind(true_par, optim_par), digits = 2) true_par optim_par X_01_conv_opt 0.8 1.1 0.4 0.5 4.0 1.9
うーん、sがちょっと外れているようですね。推定された値でのプロットを見てみましょう。
plot(dat$X_01, hill_transformation(dat$X_01, 0.4, 4), col = 2, xlim = c(0, 1), ylim = c(0, 1), ylab = "Hill Transformed Value") par(new = T) plot(dat$X_01, hill_transformation(dat$X_01, res_optim$par[1], res_optim$par[2]), col = 3, xlim = c(0, 1), ylim = c(0, 1), axes = F, ylab = "") legend("topleft", legend = c("k = 0.4, s = 4", "k = 0.49, s = 1.9"), col = c(2:3), lty = rep(1, 2))
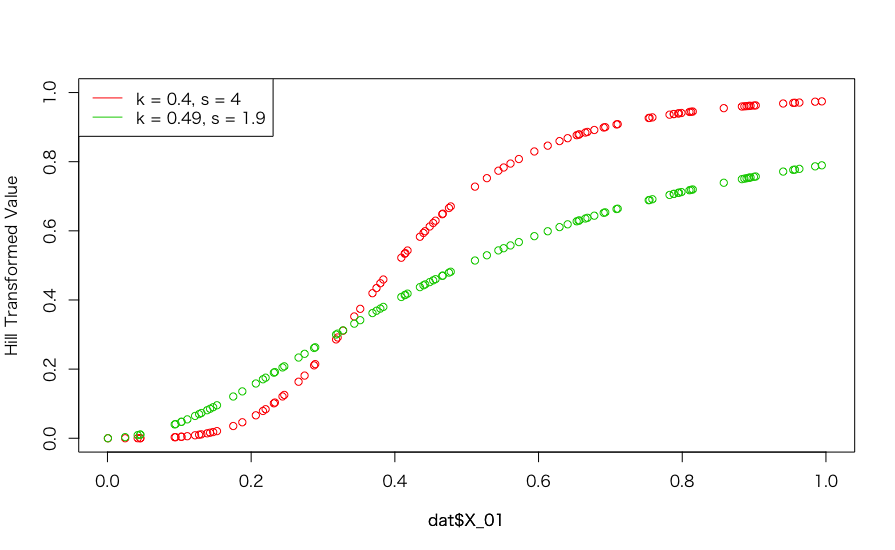
ちょっとカーブのメリハリがなく、全体として直線的になってしまっています。これが今回のサンプルで偶々発生したものなのか、それともsの推定に何らかの偏りがあるのか確かめてみましょう。同様の試行を1,000回繰り返してみます。
## 5番目のパラメータはAd-Stockのλだが今回は不使用 pars <- c(100, 5, 0.1, 0.8, 0.7, 0.4, 4) n <- 1000 res_optim_all <- matrix(NA, n, 3) param <- rep(1, 2) try_s <- function(pars) { dat <- simulate_y(pars) res_optim <- optim(par = optim(par = param, fn = return_AIC, dat = dat)$par, fn = return_AIC, dat = dat) dat$X_01_conv_opt <- hill_transformation(dat$X_01, res_optim$par[1], res_optim$par[2]) return(c(coef(lm(Y ~ X_01_conv_opt, dat))[2], res_optim$par)) } for (i in 1:n) { res_optim_all[i, ] <- tryCatch(try_s(pars), error = function(e) return(rep(0, 3))) }
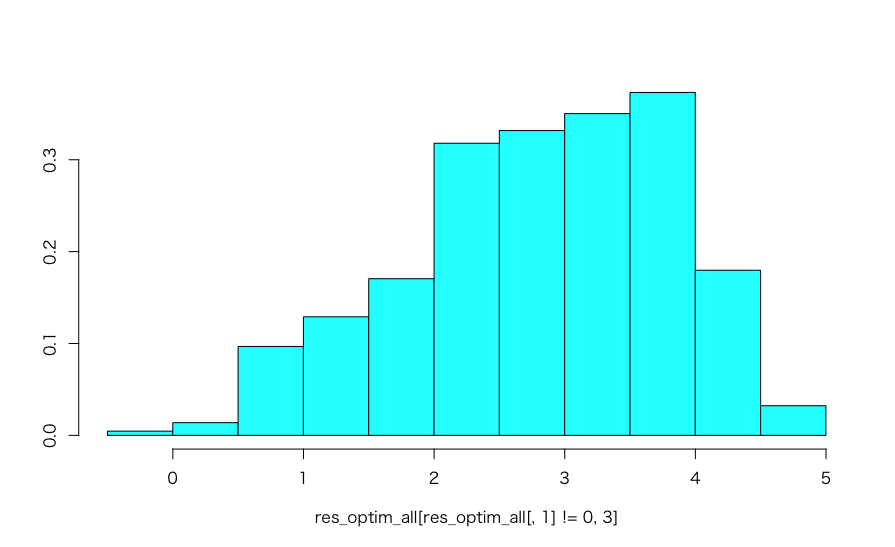
tryCatchを使って無理やりループを回しましたが、やはり推定が不安定なようで全体の4割ちょっとしか収束していません。その収束した解のバラツキを見てみても以下のようにかなり広がってしまっており、真値である4も中心とはなっていませんね。
> nrow(res_optim_all[res_optim_all[, 1] != 0.0, ]) [1] 434
MASS::truehist(res_optim_all[res_optim_all[, 1] != 0, 3])
Stanによるフィッティング
では今度はStanを使ってみましょう。以下のように指定します。
### データの再作成 set.seed(123) pars <- c(100, 5, 0.1, 0.8, 0.7, 0.4, 4) dat <- simulate_y(pars) dat_Stan <- list(N = nrow(dat), Y = dat$Y, X_01 = dat$X_01 )
またStanのスクリプトは以下のようになります。今回は特別な指定はしていませんが、X_01_convを作成するためにtransformed_parametersブロックを置いています。
data { int N; vector[N] Y; vector[N] X_01; } parameters { real mu; real beta_01; real<lower=0, upper=1> shape_k_01; real<lower=0, upper=5> shape_s_01; real<lower=0> var_error; } transformed parameters { vector[N] X_01_conv; for(k in 1:N) { X_01_conv[k] = 1 / (1 + (X_01[k] / shape_k_01)^-shape_s_01); } } model { // Sampling for(i in 1:N) { Y[i] ~ normal(mu + beta_01 * X_01_conv[i], var_error); } }
上記のモデルを用いて、フィッティングしてみましょう。
fit_01 <- stan(file = '/YourDirectory/Hill_Transformation.stan', data = dat_Stan, iter = 10000, chains = 4, seed = 123)
結果の確認
フィッティングが終わったので、結果を見てみましょう。fit_01からサンプルを抽出して加工します。
## サンプルを抽出する res_01 <- rstan::extract(fit_01) ## 該当するパラメータを取り出す ests <- summary(fit_01)$summary beta_rows <- rownames(ests)[grep("beta", rownames(ests))] shape_rows <- rownames(ests)[grep("shape", rownames(ests))]
まずは収束の判断です。Rhatが1.1未満であることを確認した上で、トレースプロットとヒストグラムを見てみましょう。
stan_trace(fit_01, pars = beta_rows) stan_trace(fit_01, pars = shape_rows) stan_hist(fit_01, pars = beta_rows) stan_hist(fit_01, pars = shape_rows)



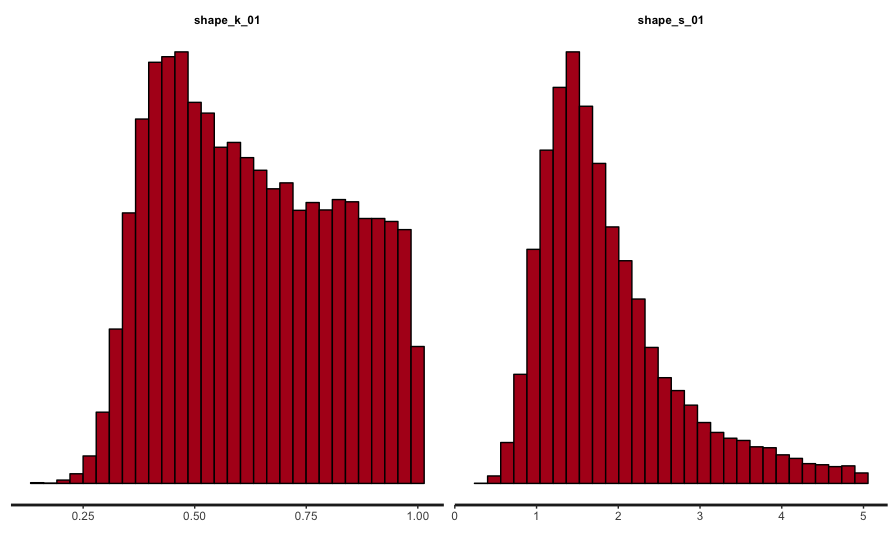
うーん、kが全体的に広がっていたり、sは真値とかけ離れたところにピークがあったり、推定に失敗している様子ですね。
ひとまず回帰係数の推定結果を見てみましょう。実際の値と推定値を並べてみます。
beta_par <- ests %>% data.frame %>% select(mean) %>% mutate("Par" = rownames(ests)) %>% filter(Par %in% beta_rows)
> sprintf("True = 0.8, Stan_Est = %.3f, Optim_Est = %.3f", beta_par$mean, optim_par[1]) [1] "True = 0.8, Stan_Est = 1.369, Optim_Est = 1.111"
あらら、optimの結果よりも外れてしまいました。Shape効果の方はどうでしょうか。
> ests %>% + data.frame() %>% + select(mean) %>% + mutate(Par = rownames(ests), + Type = "Stan") %>% + filter(Par %in% shape_rows) %>% + bind_rows(data.frame( + mean = c(0.4, 4, optim_par[-1]), + Par = rep(c("shape_k_01", "shape_s_01"), 2), + Type = c(rep("True", 2), rep("Optim", 2)) + )) %>% + spread(Type, mean) %>% + select(Par, True, Stan, Optim) Par True Stan Optim 1 shape_k_01 0.4 0.6308075 0.4965623 2 shape_s_01 4.0 1.8736434 1.9041772
こちらもoptimの方がまだ設定値に近いようですね。ただoptimもstanもどちらもうまく推定できていない様子なので比較に意味はないかもしれませんが。
終わりに
というわけで、今回はあまりパッとしない結果となってしまいました。元論文はちゃんと読んでいないのですが、彼らは階層ベイズで解いているようなので、もう少しパラメータに対して制約をかけてあげないとダメなのかもしれません。
本当なら良い推定値が得られるようになるまで頑張りたいのですが、ひとまず思いつくことは試した上でこの結果なので、いったん諦めて公開することとしました。何かアイディアが浮かべば再挑戦したいと思います。
-
要するにAd-Stock効果のパラメータの同時推定がうまく行かなかったんです。。。↩
RでWebスクレイピングしたい
背景
ちょっとした用事によりリコール情報について調査する機会がありました。これまでWebスクレイピングは経験がなかったのですが、便利なライブラリ({rvest})もあることだし、挑戦してみた結果を紹介します。 内容としては、国交省のサイトにある「リコール情報検索」(こちら)からリコールデータを取得し、テキストマイニングにかけた、というものです。
分析の進め方
分析の進め方は以下の通りです:
特別なことはしておらず、サイトのページ構成に合わせて必要なデータを取得し、可視化などを行います。
1.サイトのページ構成を把握
ここは、Rではなくブラウザの機能を使いました。例えばこの辺りの記事を参考に、Google Chromeのデベロッパーツールでhtmlの構成を把握しました。
2.構成にマッチするようにループを組んでrvest::read_htmlで順次読み込み
ライブラリのインストール
ここからがRによる処理となります。まずは必要なライブラリをインストールして読み込みます。今回新しくインストールしたライブラリは以下の通りで、RMecabはこちらの記事を参考にMecabのインストールから行いました。以下は、Mecabのインストールが終わっている前提です。
install.packages("rvest") install.packages("RMeCab", repos = "http://rmecab.jp/R") install.packages("wordcloud")
ライブラリの読み込み
インストールしたライブラリ以外に、{dplyr}や{tidyr}などの定番ライブラリ、またテキストデータを扱うので{stringr}や{stringi}なども読み込んでいます。
library(rvest) library(dplyr) library(tidyr) library(stringr) library(stringi) library(RMeCab) library(ggplot2) library(wordcloud)
{RMecab}のお試し
ここで少し{RMecab}を使ってみましょう。こんな使い方ができます。
res <- RMeCabC("すもももももももものうち") > unlist (res) 名詞 助詞 名詞 助詞 名詞 助詞 名詞 "すもも" "も" "もも" "も" "もも" "の" "うち"
{rvest}のお試し
同じく{rvest}も試してみます。read_htmlで指定したURLのページ構成をごそっと取ってきてくれます。
source_url <- "http://carinf.mlit.go.jp/jidosha/carinf/ris/search.html?selCarTp=1&lstCarNo=1060&txtMdlNm=&txtFrDat=2000/01/01&txtToDat=2017/12/31&page=1" recall_html <- read_html(source_url, encoding = "UTF-8")
取ってきたデータの中身を確認するためには、例えば以下のようにします:
> recall_html %>% + html_nodes("body") %>% # HTMLのbodyタグの情報を取り出す + html_text() # テキストデータを取り出す [1] "\n\n\n \n \n \n \n \n \n \n \n \n \n \n \n トップページ>リコール情報検索>リコール届出情報一覧\n \n \n \n \n \n \n リコール届出情報一覧\n ご利用のブラウザはJavaScriptまたはCookieが無効に設定されています。設定を確認して再度アクセスしてください。\n \n\n\n\n\n \n 90件のデータがヒットしました\n \n \n 5 / 5 ページ\n \n 番号\n 届出番号\n 届出日\n 通称名\n 81\n リ 国-3988-0\n 2017/02/02\n [リバティ]\n 82\n リ 国-3988-0\n 2017/02/02\n [ブルーバードシルフィ]\n 83\n リ 国-3988-0\n 2017/02/02\n [キャラバン]\n 84\n 改 国-0513-0\n 2017/01/27\n [デイズ]\n 85\n 改 国-0513-0\n 2017/01/27\n [デイズ ルークス]\n 86\n リ 国-3944-1\n 2017/01/27\n [デイズ]\n 87\n リ 国-3944-1\n 2017/01/27\n [デイズ ルークス]\n 88\n リ 国-3944-2\n 2017/01/27\n [デイズ]\n 89\n リ 国-3944-2\n 2017/01/27\n [デイズ ルークス]\n 90\n リ 国-3977-0\n 2017/01/27\n [セレナ]\n \n \n\n \n \n \n \n\n\t \n\n \n \n\n\n\n \n \n \n \n \n \n \n \n トップページ\n 自動車のリコール制度について\n リコール情報検索\n リコール届出情報一覧\n 自動車不具合情報ホットライン\n 不具合情報検索\n 事故・火災情報検索\n よくあるお問い合わせ\n 公表資料\n 自動車を安全に使うためには\n 利用規約等\n バナーダウンロード\n \n \n \n \n \n\n\n Copyright © 2001-myDate = new Date() ;myYear = myDate.getFullYear();document.write(myYear); Ministry of Land, Infrastructure, Transport and Tourism All rights reserved.\n\n\n\n\n(function(){\n var p = ((\"https:\" == document.location.protocol) ? \"https://\" : \"http://\"), r=Math.round(Math.random() * 10000000), rf = window.top.location.href, prf = window.top.document.referrer;\n document.write(unescape('%3C')+'img src=\"'+ p + 'acq-3pas.admatrix.jp/if/5/01/7b9f970b303989123e334299f50a384a.fs?cb=' + encodeURIComponent(r) + '&rf=' + encodeURIComponent(rf) +'&prf=' + encodeURIComponent(prf) + '\" alt=\"\" width=\"1\" height=\"1\" '+unescape('%2F%3E'));\n})();\n\n\n\n\nnew Image(1, 1).src=\"//data-dsp.ad-m.asia/dsp/api/mark/?m=323gb&c=bcMR&cb=\" + Math.floor(new Date().getTime() / 86400);\n\n (function(i,s,o,g,r,a,m){i['GoogleAnalyticsObject']=r;i[r]=i[r]||function(){\n (i[r].q=i[r].q||[]).push(arguments)},i[r].l=1*new Date();a=s.createElement(o),\n m=s.getElementsByTagName(o)[0];a.async=1;a.src=g;m.parentNode.insertBefore(a,m)\n })(window,document,'script','//www.google-analytics.com/analytics.js','ga');\n\n ga('create', 'UA-52116336-5',{'allowLinker': true});\n ga('require','linker');\n ga('linker:autoLink',['destination']);\n ga('require','displayfeatures');\n ga('send', 'pageview');\n\n"
またページの全てのtableのテキストを取り出す時はこのようになります:
> recall_html %>% + html_nodes(xpath="//table") %>% + html_text() [1] "番号\n 届出番号\n 届出日\n 通称名\n 81\n リ 国-3988-0\n 2017/02/02\n [リバティ]\n 82\n リ 国-3988-0\n 2017/02/02\n [ブルーバードシルフィ]\n 83\n リ 国-3988-0\n 2017/02/02\n [キャラバン]\n 84\n 改 国-0513-0\n 2017/01/27\n [デイズ]\n 85\n 改 国-0513-0\n 2017/01/27\n [デイズ ルークス]\n 86\n リ 国-3944-1\n 2017/01/27\n [デイズ]\n 87\n リ 国-3944-1\n 2017/01/27\n [デイズ ルークス]\n 88\n リ 国-3944-2\n 2017/01/27\n [デイズ]\n 89\n リ 国-3944-2\n 2017/01/27\n [デイズ ルークス]\n 90\n リ 国-3977-0\n 2017/01/27\n [セレナ]\n "
本番
それではここからが本番です。まずは対象となるページと、したいことが何であるかを確認しておきましょう。

こちらが今回の分析対象となるページです。この検索条件として例えば車名を「ニッサン」、届出日を「2017/01/01」〜「2017/12/31」としてみましょう。

このように条件に合致したリコール情報を一覧表示してくれます。例えば1個目をクリックすると、

リコール情報の詳細について知ることができます。このうち、上段の表にある「車名/メーカー名」や「不具合装置」、「対象台数」などを取得したいのですが、リンクを一つずつ辿ってコピーしてくるのは大変なので、スクリプトを書いて情報を取ってきたい、というのが今回の取り組みです。
スクリプトの大まかな内容としては、
- 検索結果の画面から、各リコールの詳細結果画面へのURLを取得する
- 取得したURLに順次アクセスし、必要な情報を取り出してまとめる
- 次のページに移動し、繰り返し
という感じになります。
3についてですが、幸いなことに1の検索結果URLは、次ページを確認すると末尾が「page=2」となっています。ここから元のページに戻ると「page=1」となっており、数値を変更するだけで任意のページに行けそうなので、検索結果のページ数(今回は5)だけメモしておけばループで回せそうです。 また、各リコールの詳細結果画面についてはURLが「http://carinf.mlit.go.jp/jidosha/carinf/ris/detail/1141591.html」 のようになっており、末尾の「数字7桁」を変えていけば良さそうです。
というわけで、以下のように検索結果と各リコールの詳細結果画面のURLについて、変更がない部分を定義しておきます。
src_url <- "http://carinf.mlit.go.jp/jidosha/carinf/ris/search.html?selCarTp=1&lstCarNo=1060&txtMdlNm=&txtFrDat=2017/01/01&txtToDat=2017/12/31&page=" link_url <- "http://carinf.mlit.go.jp/jidosha/carinf/ris/"
また分析に用いる項目を以下の5つとし、結果の格納用のデータフレームを準備しておきます。
target_column <- c("車名/メーカー名", "不具合装置", "状 況", "リコール開始日", "対象台数") html_tbl_all <- data_frame()
以下、リコール情報を順次取得していきます。スクリプトの流れのセクションで書いたように、検索結果の画面のページを変えつつ、各リコールの詳細結果画面へのURLを取得し、read_htmlでデータを取り出していきます。
下のスクリプトでtarget_url_listをsapplyすればもっと早くなるかもしれませんが、今回はパフォーマンスを求めたい訳ではないので素直にLoopを回しました。
st <- Sys.time() ## iはページ数。事前にメモしておく。今回は5 for (i in 1:5) { ## 検索結果の各ページのURLを指定し、データを取得 target_page <- paste0(src_url, i) recall_html <- read_html(target_page, encoding = "UTF-8") ## 検索結果画面から、各リコール詳細結果へのURLを取得 target_url_list <- recall_html %>% html_nodes(xpath = "//a") %>% # aタグに格納されている html_attr("href") %>% # href属性のデータを取り出す as_data_frame() %>% filter(grepl("detail", .$value)) # 詳細結果は"detail" + 数字7桁 + .htmlで構成されている ## 詳細結果の数 l <- nrow(target_url_list) ## ここから各詳細結果へアクセスし、データを取得する for (j in 1:l) { ## アクセス負荷を軽減するため、少し間を置く Sys.sleep(2) ## 詳細結果へのURLを指定し、データを取得 target_url <- paste0(link_url, target_url_list$value[j]) recall_html_tmp <- read_html(target_url) html_tbl_tmp <- html_table(recall_html_tmp)[[1]] ## 上段のテーブルのデータを取得 ## 4列あるが、1・3列目に項目名が、2・4列目にデータが入っているので、2列のデータに直す html_tbl <- html_tbl_tmp %>% filter(X1 %in% target_column) %>% ## 必要な情報を抽出 rename("Term" = X1, "Value" = X2) %>% select(Term, Value) %>% bind_rows( html_tbl_tmp %>% filter(X3 %in% target_column) %>% rename("Term" = X3, "Value" = X4) %>% select(Term, Value)) %>% spread(Term, Value) ## 順次追加していけるよう、wideに変換 ## データを追加 html_tbl_all <- bind_rows(html_tbl_all, html_tbl) } }
> Sys.time() - st Time difference of 9.495965 mins
このスクリプトでは一年分の日産のデータを取得するのに、私の環境で約10分かかりました。結構時間がかかるので、データを保存しておきます。
save(html_tbl_all, file = "Recall_Data.Rdata")
3.取得したテキストデータをMecabで形態素解析
ではこれ以降、取得したデータで分析を行います。と言ってもMecabによる形態素解析を掛けた後は集計して可視化するぐらいのものです。その前にデータを確認してみましょう。検索結果では90件と表示されていましたが、ちゃんと取れているでしょうか。
> dim(html_tbl_all) [1] 90 5
大丈夫なようですね。データも見てみましょう。
> head(html_tbl_all) # A tibble: 6 x 6 リコール開始日 `車名/メーカー名` `状 況` 対象台数 不具合装置 Num <chr> <chr> <chr> <chr> <chr> <dbl> 1 2017年12月14日 いすゞ ① 電源電圧が12Vのテールゲートリフタ装着車の後部反射器において、選定が不適切なため、反射… 1,153台 その他(保安灯火)… 1153 2 2017年12月14日 いすゞ ② 電源電圧が24Vのテールゲートリフタ装着車の後部反射器及び後退灯において、選定が不適切な… 162台 後退灯 162 3 2017年12月15日 ニッサン 電源分配器の基板において、回路基板の製造時に不要な半田が付着した状態で防湿材がコーティングさ… 316,759… その他(電気装置)… 316759 4 2017年12月15日 ニッサン 電源分配器の基板において、回路基板の製造時に不要な半田が付着した状態で防湿材がコーティングさ… 316,759… その他(電気装置)… 316759 5 2017年12月15日 ニッサン 電源分配器の基板において、回路基板の製造時に不要な半田が付着した状態で防湿材がコーティングさ… 316,759… その他(電気装置)… 316759 6 2017年12月01日 いすゞ 小型トラックの燃料噴射装置において、サプライポンプをエンジンに締結する取付けボルトの締付トル… 83,591台 エンジン一般… 83591
「車名」を確認すると一部にニッサン以外が含まれていますね。しかし当該の詳細結果を確認すると「いすゞ」とともに「ニッサン」がリコール対象となっており、間違いではないようです。
また「状況」を確認すると、全く同じ文言が同じ日付で出ています。これは、このリコールの届出が車種別に行われているためであり、例えば「2017年12月15日」の例では、「セレナ」「キューブ」「バネット」で同じ理由によりリコールの届出があったようです。本来この後の形態素解析では、これらのテキストを集約するべきでしょう(今回はお試しなのでやりませんが)。
分析対象となる部分を取り出す
さて、今回形態素解析の対象としたいテキストデータは「状 況」です。RMecabではテキストファイルからデータを読み込んで処理するので、テキストとして書き出しておきましょう。
load("Recall_Data.Rdata") ## 必要なら txt_defect_situation <- html_tbl_all$`状 況` write.csv(txt_defect_situation, file = "Situation.csv")
読み込み
書き出したテキストファイルを以下のように読み込みます。
txt_situ <- RMeCabFreq("Situation.csv")
全部で529個の単語に分割されたようです。
データ加工
テキストデータはMecabによって形態素解析され、単語ごとに分割された上で品詞を割り当てられています。このうち、単語抽出の対象となりそうなものだけを使用します。今回は名詞の頻度を確認します。
Noun_res_situ <- txt_situ %>% filter(Info1 == "名詞") %>% filter(!Info2 %in% c("非自立", "代名詞")) %>% group_by(Term, Info1) %>% summarise("TF" = sum(Freq)) %>% ungroup() %>% arrange(desc(TF)) %>% mutate(Pos = factor(Term, levels = .$Term))
上のスクリプトで最後にfactorにしているのは、グラフにする時に単語の並び順ではなく頻度で表示するためです。
4.可視化
では加工済みのデータを用いて単語の頻度を可視化してみましょう。まずは棒グラフですが、単語の数が多いので上位20個に限定しています。なおMacの場合、日本語が表示されない可能性があります(私は表示されませんでした)。その場合、下記のページが参考になると思います。私は下記を全て実行したところ表示できるようになりました:
- http://blog.0093.tv/2011/05/rstudio-for-mac-os-x.html
- https://ameblo.jp/mojio914/entry-12030044452.html
- https://www.karada-good.net/analyticsr/r-58
ggplot(Noun_res_situ[1:20, ], aes(x = Pos, y = TF)) + geom_bar(stat = "Identity") + theme_bw(base_family = "HiraKakuProN-W3")

「"」や「","」のような変な単語(?)が混ざっていますね。これは流石に格好悪いので除いておきます。
Noun_res_situ %>% filter(!Term %in% c("\"", "\",\"")) %>% slice(1:20) %>% ggplot(., aes(x = Pos, y = TF)) + geom_bar(stat = "Identity") + theme_classic(base_family = "HiraKakuProN-W3")

なるほど、なんかそれっぽい単語が抽出されていますね〜。しかし、実際のテキストを見ていると、例えば「検査」という単語は「完成検査」という熟語として使われることが多いなど、ドメイン特有の表現があったりします。そういった特有の表現を集めた辞書がないと、こういった単語抽出はあまり効果的でなかったりします。
続いてWordcloudを作成してみます。ここでも単語の数が多いので絞ろうと思うのですが、個数ではなく出現頻度でfilterしましょう。TFが4以上の単語を抽出すると、以下のようになります。
Noun_res_situ_4 <- Noun_res_situ %>% filter(!Term %in% c("\"", "\",\"")) %>% filter(TF >= 4) par(family = "HiraKakuProN-W3") wordcloud(Noun_res_situ_4$Term, Noun_res_situ_4$TF, random.color = TRUE, colors = rainbow(10))

5.おまけ
以上でやりたかったことは終わりなのですが、最後におまけでリコール総台数の確認をしてみます。「対象台数」列に入っている数値を集計したいのですが、文字列として入力されているので修正します。
html_tbl_all$Num <- html_tbl_all$対象台数 html_tbl_all$Num <- str_replace_all(html_tbl_all$Num, ",", "") html_tbl_all$Num <- str_replace_all(html_tbl_all$Num, "台", "") html_tbl_all$Num <- as.numeric(html_tbl_all$Num) ggplot(html_tbl_all, aes(x = Num)) + geom_histogram() + theme_classic()

最後に
というわけで今回は{rvest}を用いたWebスクレイピングに挑戦しました。read_htmlで簡単にWebサイトのデータを取得することができ、html_text()やhtml_table()で簡単に加工することが可能なため、初めての挑戦ではあったものの大きな引っかかりもなく進めることができました。今まで何となく敬遠していたのですが、積極的に使っていきたい技術ですね。
階層ベイズと状態空間モデルで広告効果を推定したい
背景
これまでMarketing Mix Modeling(MMM)におけるAdStock効果の推定について色々と記事を書いてきましたが、その他にも試したいと思っているモデルがいくつかあります。その一つが階層ベイズモデルと状態空間モデルを同時に取り扱うものです。
例えば「地域別の売上推移のデータ」が手元にあると考えてみましょう。地域ではなく人や商品でも構いませんが、ある要因の各水準がそれぞれ時系列データを持っている状況(いわゆるパネルデータ)で、ひとまずここでは地域とします。このようなデータはあらゆる会社で保有していることでしょう。 今、各地域についてMMMにより広告効果を推定することを考えたとき、どのようなモデリングが可能でしょうか?
シンプルに考えれば、地域ごとに一つずつモデルを作るという方法が挙げられます。例えば地域の数が2つ3つしかなかったり、モデルの作成に時間をかけることが可能であればこの方法は有効かもしれません。しかし問題もあります。地域ごとに独立してモデルを作成するということは、各地域のパラメータは互いに独立であるとの仮定を置くことになるのですが、それは事実でしょうか?
確かにTVや新聞には地方欄やローカル局があり、地域限定のコンテンツや提供される場合もあるでしょう。しかしどちらかと言えば全国で同じコンテンツが提供される割合の方が大きいでしょうし、購買行動に地域による差がそれほど認められるとは、経験的にも思えません。もし仮に地域による異質性というものがそれほど強くないのであれば、むしろそういった情報を積極的に活かした方が良いのではないでしょうか?
目的
そのような時に、パラメータ間に緩やかな縛りをかける方法として階層ベイズモデルという手法があります。詳しくは書籍1を参考にして頂くとして、ここでは「各地域の広告効果を表すパラメータを生成する分布を仮定する」方法と説明しておきます。この分布の幅(バラつき、分散)が十分に大きければ広告効果は地域によって様々な値を取り得ますし、逆に分布の幅が極端に狭ければ地域ごとの広告効果に差はないことを意味します。
この階層構造を持つモデルを、時系列データの分析でお馴染みの状態空間モデルと合わせて取り扱いたいというのが今回の試みです。具体的には、以下のようなモデルを想定しています。
添字のAおよびtはそれぞれ地域(Area)と時点(time)を意味しており、は「ある地域
Aのt時点における観測値」となります。
ポイントは4行目で、地域ごとの広告効果に対して、その生成元となる分布(ここでは正規分布)を仮定しています。もし
が大きければ
は様々な値を取り得ますが、値が小さければ非常に似通った値になることが理解できると思います。
ライブラリの読み込み
今回の分析では{rstan}を使用します。階層ベイズ + 状態空間モデルといった非常に複雑なモデルを表現できるのが強みです。
library(tidyverse) library(ggplot2) library(rstan) options(mc.cores = parallel::detectCores()) rstan_options(auto_write = TRUE)
シミュレーションデータの生成
分析用のデータを生成しますが、検証のポイントとしては以下のようになります:
- 5地域(
num_region)から4年分(num_year)の月次(num_month)データを収集し、合計240点の観測値を得たと想定 - 回帰係数(広告効果)は平均
0.5、分散0.01の正規分布にしたがって発生 - 状態変数のパラメータは各地域で共通
## シードの固定 set.seed(123) ## 観測値の数 num_region <- 5 num_month <- 12 num_year <- 4 data_length <- num_month * num_year ## 回帰係数 mu_beta <- 0.5 var_beta <- 0.01 beta_ad <- rnorm(num_region, mu_beta, sqrt(var_beta)) beta_ad_all <- rep(beta_ad, each = data_length) ## 分布の設定 # 状態変数の初期値 state_t0 <- 3 var_state_t0 <- 1 # 状態変数の分散 var_state <- 0.01 # 誤差項 var_error <- 0.01 ## 説明変数X scale_x <- 5 zero_per <- 0.2 X <- ifelse(runif(num_region * data_length) > zero_per, 1, 0) * rpois(num_region * data_length, scale_x)
上記の条件に従いデータを発生させます。状態変数を除けば先に示したモデルの通り簡単な線形モデルです。Area_IDは、後ほどStanでフィッティングを行うために必要となる変数で、YMは描画用です。
## 状態変数 State <- matrix(0, nrow = num_region * data_length) for (i in 1:num_region) { for (j in 1:data_length) { if (j == 1) { ## 1, 49, 97, 145, 193行目が各地域の先頭となる State[(i-1) * data_length + j] <- rnorm(1, state_t0, sqrt(var_state_t0)) } else { State[(i-1) * data_length + j] <- State[(i-1) * data_length + j - 1] + rnorm(1, 0, sqrt(var_state)) } } } ## 目的変数 error <- rnorm(num_region * data_length, 0, sqrt(var_error)) Y <- State + X * beta_ad_all + error ## 地域ごとのID Area_ID <- 1:num_region DF <- data.frame( "Area_ID" = rep(Area_ID, each = data_length), "YM" = rep(1:data_length, time = num_region), "Y" = Y, "X" = X, "True_s" = State, "True_e" = error)
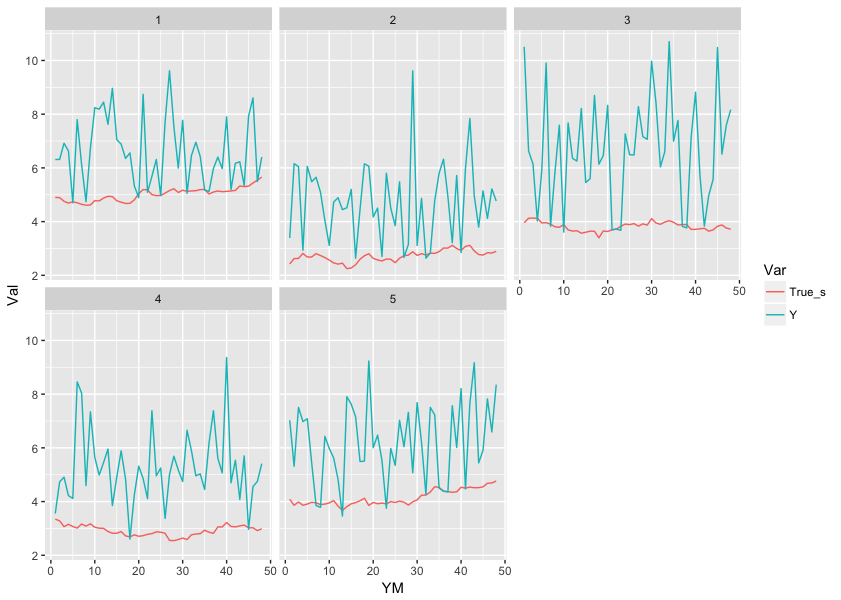
ここで観測値と状態変数が各地域でどのように推移しているか確認しておきます。
DF %>% gather("Var", "Val", -c(Area_ID, YM)) %>% filter(Var %in% c("Y", "True_s")) %>% ggplot(., aes(x = YM, y = Val, color = Var)) + geom_line() + facet_wrap(~Area_ID)
また、この時点で地域をまとめてlmで推定した$\beta$がどのようになるかも確認しておきましょう。
> coef(lm(Y ~ X, data = DF)) (Intercept) X 3.8088184 0.5003114
また地域ごとにデータを分割した場合も試してみます。
results <- list() for (i in Area_ID) { results[[as.character(i)]] <- coef(lm(Y ~ X, data = DF, subset = Area_ID == i)) }
> print(cbind(do.call("rbind", results), beta_ad)) (Intercept) X beta_ad 1 5.066906 0.4292258 0.4439524 2 2.700195 0.4837768 0.4769823 3 3.764447 0.6628493 0.6558708 4 2.953617 0.4988667 0.5070508 5 4.051693 0.5314325 0.5129288
そこそこ近い値が推定されていますね(汗
Stanによるフィッティング
気を取り直して、Stanを用いたフィッティングを実行してみます。Stanに渡すデータは以下の通りです。
dat_Stan <- list(N = data_length, K = num_region, Y = DF$Y, X = DF$X, Area_ID = DF$Area_ID)
またStanのスクリプトは以下のようになります。
data { int N; // 地域ごとの観測値の数(data_length) int K; // 地域の数(num_region) vector[N*K] Y; // 観測値のベクトル vector[N*K] X; // 説明変数のベクトル int<lower=1, upper=K> Area_ID[N*K]; } parameters { real state_t0; // 状態変数の0期目の平均 vector[N*K] state; // 状態変数のベクトル real beta_0; // 回帰係数の事前分布の平均 vector[K] beta; // 地域ごとの回帰係数のベクトル real<lower=0> var_state_t0; // 状態変数の0期目の分散 real<lower=0> var_state; // 状態変数の分散 real<lower=0> var_beta_0; // 回帰係数の事前分布の分散 real<lower=0> var_error; // 誤差分散 } model { // 状態変数をサンプリング for (k in 1:K) { // 1期目の値は0期目の分布からサンプルする state[1 + (k-1)*N] ~ normal(state_t0, var_state_t0); // 2期目以降は前期の値を平均とした分布からサンプルする for(i in 2:N) { state[i + (k-1)*N] ~ normal(state[i-1 + (k-1)*N], var_state); } } // 回帰係数をサンプリング for (k in 1:K) { beta[k] ~ normal(beta_0, var_beta_0); } // Yをサンプリング for(i in 1:(N*K)) { Y[i] ~ normal(state[i] + beta[Area_ID[i]] * X[i], var_error); } }
このスクリプトをHB_SSM_Sim.stanとして保存し、フィッティングを行います。
fit_01 <- stan(file = '/YourDirectory/HB_SSM_Sim.stan', data = dat_Stan, iter = 1000, chains = 4, seed = 123)
結果の確認
フィッティングが終わったので、結果を見てみましょう。fit_01からサンプルを抽出して加工します。
## サンプルを抽出する res_01 <- rstan::extract(fit_01) ## 該当するパラメータを取り出す ests <- summary(fit_01)$summary t0_rows <- rownames(ests)[grep("state_t0", rownames(ests))] state_rows <- rownames(ests)[grep("state\\[", rownames(ests))] b0_rows <- rownames(ests)[grep("beta_0", rownames(ests))] beta_rows <- rownames(ests)[grep("beta\\[", rownames(ests))] ## 状態変数 state_par <- ests %>% data.frame %>% select(mean) %>% mutate("Par" = rownames(ests)) %>% filter(Par %in% state_rows) %>% mutate("Area" = DF$Area_ID) state_cmp <- data.frame( True = State, Est = state_par$mean, Area = as.factor(state_par$Area), YM = DF$YM )
まずは状態変数の推定結果を見てみましょう。実際の値と推定値を並べてみます。 (なお以降の作業の前にサンプルのtraceプロットとヒストグラムを確認し、収束していると判断しています)
state_cmp %>% gather("Var", "Val", -c(Area, YM)) %>% ggplot(., aes(x = YM, y = Val, colour = Var)) + geom_line() + facet_wrap(~Area)
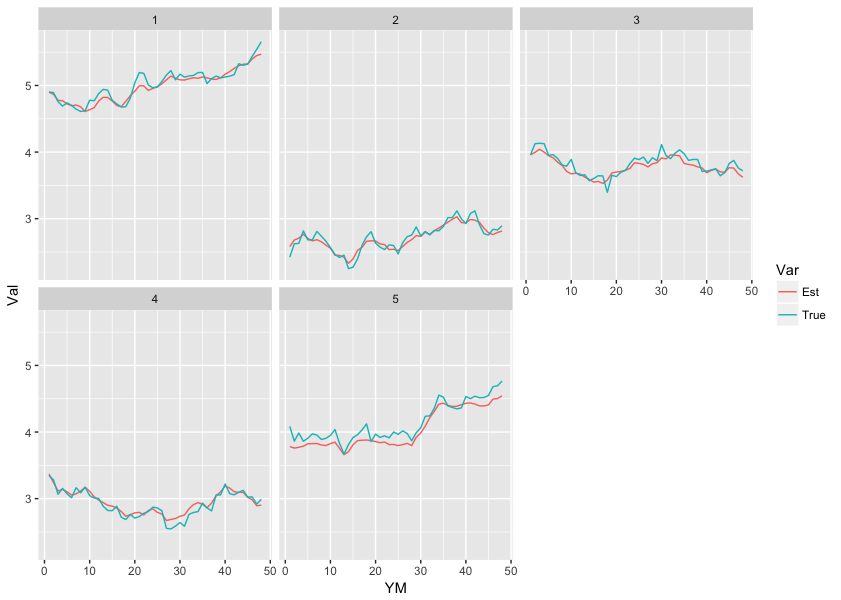
おぉ、かなり精度良く推定できているようですね!状態変数の初期値や分散の推定値はどうでしょうか?
> ests %>% data.frame %>% select(mean) %>% rename("Estimated" = mean) %>% mutate("Par" = rownames(ests)) %>% filter(Par %in% t0_rows) %>% mutate("True" = c(state_t0, var_state_t0)) %>% mutate("Simulated" = c( mean(DF[c(1, 49, 97, 145, 193), "True_s"]), var(DF[c(1, 49, 97, 145, 193), "True_s"]) )) %>% select(Par, True, Simulated, Estimated) Par True Simulated Estimated 1 state_t0 3 3.7429556 3.731479 2 var_state_t0 1 0.8521877 1.328192
状態変数初期値の平均および分散の真の値がそれぞれ3および1であったのに対し、生成されたデータでは3.7および0.85でした。推定された値は3.7および1.3で、分散がやや過大に推定されているようです。ただし以下に示すように、推定値の95%信用区間も結構広いため、逸脱しているとまでは言えないようです。
> ests %>% data.frame %>% select(X2.5., X97.5.) %>% rename("Lower95" = X2.5., "Upeer95" = X97.5.) %>% mutate("Par" = rownames(ests)) %>% filter(Par %in% t0_rows) %>% select(Par, everything()) Par Lower95 Upeer95 1 state_t0 2.3975622 5.027106 2 var_state_t0 0.5549254 3.525860
続いて回帰係数はどうでしょうか。
beta_par <- ests %>% data.frame %>% select(mean) %>% mutate("Par" = rownames(ests)) %>% filter(Par %in% beta_rows) beta_cmp <- data.frame( True = beta_ad, Est = beta_par$mean ) ggplot(beta_cmp, aes(x = True, y = Est)) + geom_point() + coord_fixed()
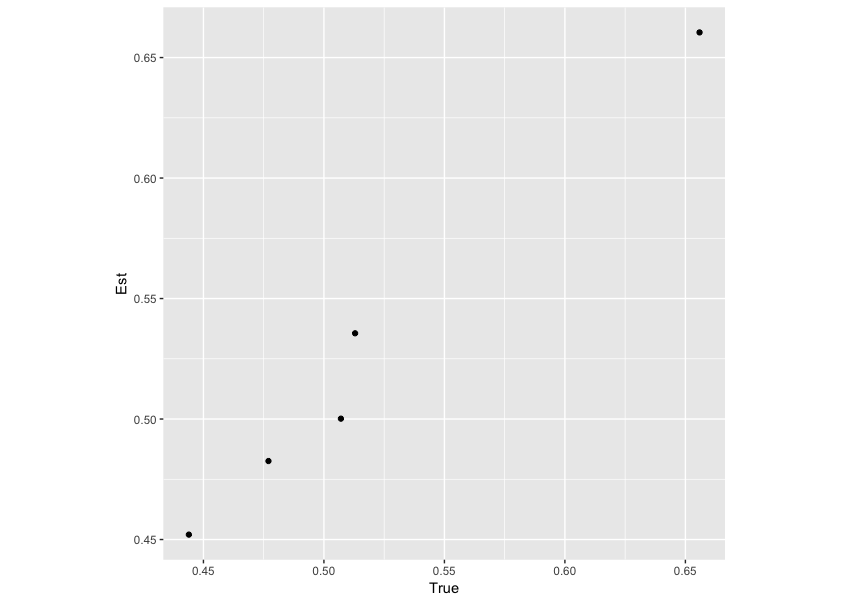
こちらも、事前に設定した回帰係数と推定値が似通っているようです。数値を確認しても、良い精度で推定できています。
ests %>% data.frame %>% select(mean) %>% mutate("Par" = rownames(ests)) %>% filter(Par %in% beta_rows) %>% bind_cols("True" = beta_ad) %>% select(Par, True, mean) Par True mean 1 beta[1] 0.4439524 0.4520343 2 beta[2] 0.4769823 0.4825661 3 beta[3] 0.6558708 0.6604340 4 beta[4] 0.5070508 0.5001494 5 beta[5] 0.5129288 0.5355829
最後に、回帰係数の事前分布についても見ておきましょう。
> ests %>% data.frame %>% select(mean) %>% mutate("Par" = rownames(ests)) %>% filter(Par %in% b0_rows) %>% bind_cols("True" = c(mu_beta, var_beta)) %>% select(Par, True, mean) Par True mean 1 beta_0 0.50 0.5184981 2 var_beta_0 0.01 0.1385978
回帰係数の事前分布の分散はちょっと大きく推定されているようですが、平均は近しい値となっているようです。
終わりに
以上、「階層ベイズと状態空間モデルを合わせて取り扱いたい」という試みでしたが、結果としては概ね満足の行くものになったと思います。序盤に書いた通り、階層ベイズ + 状態空間モデルのような非常に複雑なモデルであっても、stanを使えば非常に簡単に推定が可能です。
これまで広告効果の推定に関していくつか記事を書いてきましたが、実はゴールとなるモデルとしてはこれを想定していました。あとはこのモデルに、これまで紹介してきたようなAdStock効果の推定を組み込めば試してみたかったモデルは一通り完了することになります。
これらについても追って紹介したいと思います。
スタイン推定量を確かめてみる
いきなりですが、最近購入したベイズモデリングの世界を読んでいて非常に面白い話題を発見しました。

- 作者: 伊庭幸人
- 出版社/メーカー: 岩波書店
- 発売日: 2018/01/18
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (4件) を見る
P118から始まるスタイン推定量についてなのですが、その直感的な意味について本文から引用すると:
それぞれの
を、全部の{
}の平均値
の方向に
だけ引っ張ってやる
ことで、そのものを推定量とするよりも、パラメータの真値
との二乗誤差の期待値が小さくなるそうです。これはいわゆる縮小推定量の一種で、引っ張りの程度
をデータから適応的に求めるのがこの推定量のキモなのだそうです。
スタイン推定量の定義自体はそれほど難しいものでもなく、以下のような式によって表されます:
ここで、
です。この記事では、スタイン推定量が本当によりも二乗誤差が小さくなるのか、以下のように実験してみます。
実験内容
内容としては非常に簡単なもので、平均と分散が共に既知である正規分布からいくつかのサンプルを生成します。そのサンプルをそのまま用いた場合と、スタイン推定量を用いた場合とで、真のパラメータとの二乗誤差の期待値:
]
にどれほど差が生じるかを確認しています。
パラメータ設定
実験では、一回の試行につき平均が{1, 2, ..., n}、分散が1である正規分布に従うサンプルをn個生成することとしました。このn個のデータから各サンプルのスタイン推定量を求め、真値である{1, 2, ..., n}との二乗誤差を計算します。同時にデータそのものを用いた場合の二乗誤差も計算しておきます。上記の実験を1000回繰り返し、最終的にどちらが小さい値となるかを比較しました。
n <- 10 mu <- 1:n sig <- 1 K <- 1000 res <- matrix(0, K, 2)
実験開始
上記のパラメータに基づき、以下のように計算を行います。
set.seed(123) for (i in 1:K) { ## サンプルを発生させる tmp <- rnorm(n, mu, sig) ## スタイン推定値を求める s2 <- (1/(n-3)) * sum((tmp - mean(tmp))^2) a <- sig / s2 s_i <- (1-a)*tmp + (a/n)*sum(tmp) ## 結果を保存する res[i, 1] <- sum((s_i - mu)^2)/n # スタイン推定値を用いた二乗誤差の期待値 res[i, 2] <- sum((tmp - mu)^2)/n # データそのものを用いた二条誤差の期待値 }
結果
それでは結果です。
> mean(res[, 1] / res[, 2]) [1] 0.9618857 > sum(res[, 1] > res[, 2]) [1] 310
スタイン推定量を用いた場合、データそのものの場合と比較して二乗誤差が96%と小さくなっていることが確認できます。
終わりに
今回の実験は以上で終了です。ではこれの何が面白いのかと言うと、スタイン推定量と階層ベイズモデルとの関連性についてなんですね。現在、別に書こうとしている記事で階層ベイズモデルに触れているのですが、その説明として
パラメータを生成する分布を仮定することで、パラメータに緩やかな縛りをかける
といった言い回しを考えています。ここでふと「階層ベイズでは(緩やかな縛りをかけることで)推定値にバイアスを与えているわけだが、それにも関わらずモデルとして良くなるというのは、一体どういう意味なのだろう?」という疑問が浮かんできました。バイアスを与えているわけなのだから、誤差が大きくなってモデルとしては劣化しそうな気がしませんか?
その時にこのスタイン推定量のことを思い出しました。つまり、階層ベイズではパラメータに対する分布を仮定することで推定値にバイアスを与えているが、スタイン推定量の性質を以って全体的には誤差を小さくすることができているということではないかと1。
書籍にはスタイン推定量が誤差を減少させる証明も付いていますが、まだ読み込めていませんので上記の理解が間違っているかもしれません。しかしこの推定量自体も非常に面白く、紹介したいと思ったので実験してみた次第です。